segment里边形成的mco*.nii是配准的平均像还是啥意思
老师,请问segment里边形成的mco*.nii是配准的平均像还是啥意思,我试了一下,normalize用的的是这个文件,非常感谢
- Read more about segment里边形成的mco*.nii是配准的平均像还是啥意思
- 5 comments
- Log in or register to post comments
- 11351 reads
老师,请问segment里边形成的mco*.nii是配准的平均像还是啥意思,我试了一下,normalize用的的是这个文件,非常感谢
PicturesForChkNomalization文件夹的图片怎么判断好与不好,请老师告诉我,非常感谢
各位老师好!
我想请教一个问题:在进行reho值的组间比较时,我想把被试的结构像作为协变量加入统计,选取的是每名被试的灰质密度(通过VBM分析得到的,进行了normalize的结果)。但是因为功能像和结构像的dimension不同(功能像是61*73*61,结构像是121*145*121),REST会因此而提示无法运算。请问这一问题该如何解决?
我试了一下REST里的reslice image功能,发现如果把结构图像的voxel size改为3*3*3,得到的结果就将dimension变为61*73*61了,当然图像分辨率也变为3*3*3。将这个图像作为组间比较的协变量,这种做法正确吗?还是应该用其他方法?
请老师指教。
谢谢!
What's the content of constant and quadratic removed?Is the constant same as the Global signal?
Look forward to your reply!
老师您好,怎么理解功能像与结构像是线性相关的平动与转动,而不是扭曲的?
l'm a new learner ,why my file of RealignParameter doesn't contain the file .ps.The versions of MATLAB l try include 2009a,2012a and 2014a.Is anything wrong?
l'm a new learner ,l want to know how to open hdr file and what's the files of hdr and nill.
Thank you very much if the teacher can tell me!!!
老师您好,我现在使用的一笔数据是一个multi-site 的数据。 由于不同的site 采集fMRI时候的TR不同,所以我在使用rest toolbox计算fALFF的时候根据每个site提供了不同的TR。但是我发现汉书fALFF在计算不同TR的数据时候会对结果形成system 的bias。 比如说,一个site TR是3s,另一个site TR 是2s,那么用toolbox计算出来的fALFF就会有显著性的系统性的差异,请问这是什么原因呢,这个toolbox不适合用在muti-site的数据么?
谢谢
泽宁 傅
各位老师,我在使用DPABI做标准化时使用的Normalize+DARTEL,发现标准化后的图像匹配的并不是很好,重新调整跑了好几次,总是发现标准化后的图样跳的很离谱(如图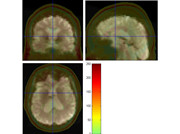 ),但是用EPI templates就不会出现这个现象,不知道是什么原因怎么解决?
),但是用EPI templates就不会出现这个现象,不知道是什么原因怎么解决?
老师,您好。我在用DPARSF4.1 ADVANCE版进行ALFF的分析,ALFF分析完之后进行了滤波。我有几点疑惑:
1. Filter这一步是对Results中ALFF/mALFF/zALFF/fALFF/fmALFF/fzALFF中的哪一个文件进行操作的?
2. 在做所有被试的ALFF或者fALFF统计的时候应该用哪个文件?是Filter之后生成的Filtered_4DVolume文件还是Results中的某一个文件?
谢谢!