老师们好:
在学习了course之后我有几个小问题想请教一下各位老师
1.关于单样本T检验:用一组病人的mReHo做单样本T检验结果(下图一)与用smReHo做出的结果(下图二)有不同,这样的话可能会导致两个样本T检验时设置两组病人都有显著变化的的并集mask时出现差异,从而影响最后结果,我想请教一下的是我们做单样本T检验时是用mReHo好呢还是用smReHo好?
图一
.bmp)
图二
2.关于报表时的BA分区书写:在报表时一个cluster我用肉眼观察发现它横跨了两个BA分区,但是用cluster report找到该cluster的peak点MNI坐标所对应的BA分区只有一个,那么我报该cluster的BA分区是否应该根据peak点对应的那一个来报?
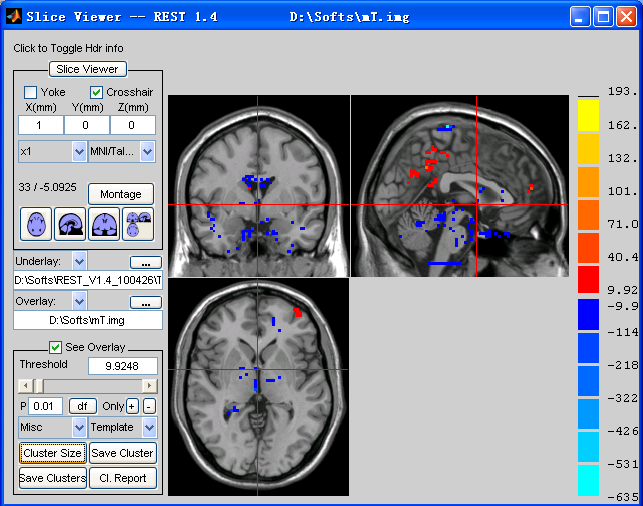
3.关于用Rest slice viewer做alphasim校正,校正前P<0.01校正之后图像上显著改变的区域倒是发生了变化,但是slice viewer显示的p值为什么还是p<0.01呢?进行多重比较校正后p值不是应该发生变化吗?
4.关于用Rest做双样本T检验时,我想要去除灰质协变量,这时我该利用DPARSF的nomalize功能分割出的wc1开头的灰质含量做协变量呢还是用mwc1开头的灰质体积做协变量呢?
5.在Rest自带的brain mask中它们命名为05 61*73*61,其中05的意思是百分之五十的阈值,而脑脊液是 07 61*73*61。这个阈值指的是什么,为什么有的选百分之五十有的选百分之七十呢?
6.若将年龄作为协变量去除,可否根据被试顺序,按男性=1,女性=0,作出一个TXT.文件作为协变量加入?
有些概念我还十分模糊,非常期盼老师们能对这几个小问题做出回答,非常感谢!!

Submitted by ZangYF on Sat, 08/14/2010 - 09:12 Permalink
首先谢谢你的问题。 1. voxel-by-voxel
Submitted by demonpupil on Sat, 08/14/2010 - 10:32 Permalink
非常感谢臧老师百忙中给予的回答! 当时我在做单样本T检验的
非常感谢臧老师百忙中给予的回答!
当时我在做单样本T检验的时候,鉴于新版REST加入了和“1”进行统计的方法后,我便利用smReho值和1进行了比较,这样做是否不妥?
Submitted by ZangYF on Sat, 08/14/2010 - 23:00 Permalink
smReHo与1进行比较,应该是对的。有多少被试?但不管怎
smReHo与1进行比较,应该是对的。有多少被试?但不管怎样,t值9对应p=0.01,并且t值达到几百,肯定是有问题的。
Submitted by demonpupil on Sun, 08/15/2010 - 11:11 Permalink
一共是三个被试
臧老师好:
一共是三个被试,数据采用的是今年去成都培训的人回来提供的数据。
Submitted by ZangYF on Sun, 08/15/2010 - 23:09 Permalink
三个样本,做统计检验几乎没什么意义。不过,看起来你的数据处
三个样本,做统计检验几乎没什么意义。不过,看起来你的数据处理过程好像没什么问题。