Submitted by vivi on Sat, 11/13/2010 - 06:43
我使用dparsf进行ALFF的处理,跑完程序之后发现11个被试中有两个的分割结果不好,灰白质和脑脊液没有分离开,是模糊的,再看他们的功能像配准也有问题。由于之前做过VBM分析,当时是直接用spm分割的,相同的被试分割效果很好(详见下图)。回想起来,中间不同的是进行resting分析的时候多了结构像和功能像coregister这一步,想请教一下为什么这一步会带来这么大的变化呢,对于这两个被试resting图像的配准有什么其他的处理办法吗(如果还是想用分割配准)。
C1 with coregisteration c2 with coregisteration c1 without coregisteration c2 without coregisteration
.png)
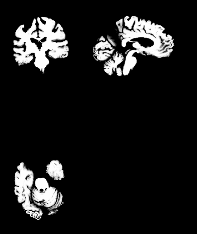
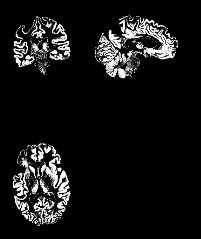
谢谢!

Submitted by ZangYF on Sat, 11/13/2010 - 15:11 Permalink
从提供的图来看,首先结果就有问题,这与coregister
从提供的图来看,首先结果就有问题,这与coregister本身关系应该不大,但需要仔细核对coregister过程,比如target与source?coregister效果如何?
Submitted by vivi on Sun, 11/14/2010 - 06:40 Permalink
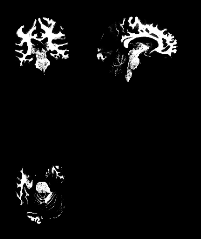
谢谢臧老师回复! 上面经过coregister的分割图是直
谢谢臧老师回复!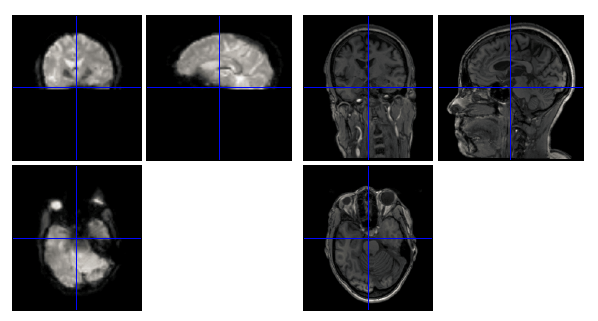
上面经过coregister的分割图是直接从dparsf处理过后的T1ImgSegment文件夹中copy出来的。我之后又参照视频中的讲解,手动用spm跑了一遍,coregister过程中,target是mean*.img,source是T1.img。然后再进行分割,出来的结果和上图是一样的。下面是coregister出来的图。
Submitted by YAN Chao-Gan on Sun, 11/14/2010 - 15:25 Permalink
Re
Hi!
一般来说coregister是不会影响分割效果的。
在DPARSF中,coregister只是改一下头文件中的affine矩阵,使得让结构像的朝向与功能向一致,而不修改数据文件本身的。
一个可能的原因是你的功能像朝向太偏,导致后面unified segmentation不能很好的进行。
你可以试一下DPARSF Advanced Edition中的Reoreient Interactively功能,在Coregister之后,再手动把图像摆正一些,有更好的分割效果。具体做法见:http://www.restfmri.net/forum/Course
Submitted by vivi on Wed, 11/24/2010 - 07:25 Permalink
Re
Thanks a lot! I've tried the reorient function. The results are pretty good!
Cheers!
Submitted by YAN Chao-Gan on Wed, 11/24/2010 - 11:38 Permalink
Re
Hi!
Thanks for your report!
Acctually, the module of "Reoreient Interactively" is designed for this situation. If you the images has a bad orientation, you can use this module before normalization or segmentation.