如题所示,我完全是这方面的小白,现在想要提取一些参数作为区分AD和NL,我手上只有结构型的MRI,所以用spm,rest这类工具有点茫然
现在我先是 通过VBM预处理,就是按照范丰梅写的那个《VBM处理流程-PDF》来的,经过了normalizing和segmenting,其中有的部分因为spm8和spm5有不同,所以我除了针对AD的预处理是thorough cleanup之外其他都是默认选项,比如那个high-demensional DATREL模板,最后使用了FWHM[8 8 8]的高斯平滑。
然后我选择了smwrp1*.nii这些文件用spm进行了双样本t检验,操作仍是参照范丰梅写的那个《VBM处理流程-PDF》.
但是有个问题,如果我使用了FDR 0.05校正,那基本上所有激活簇就没有了。。。。。所以我直接是none。
问题1:然后我用rest 的 slice viewer打开。。。。但是它弹出了这个:
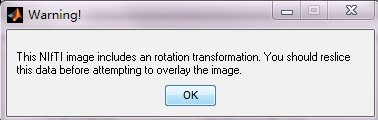
可是我查过不是说经过spm常规处理的一般不会有这个问题么????
问题2:接下来我在查看窗口里,使用的是aal模板,在这里我经过了FDR0.05校正,不知道这里的校正有什么意义呢???
问题3:如图所示,是正负激活的结果,想请问一下,这里面超出模板范围的这些激活区域怎么办呢???
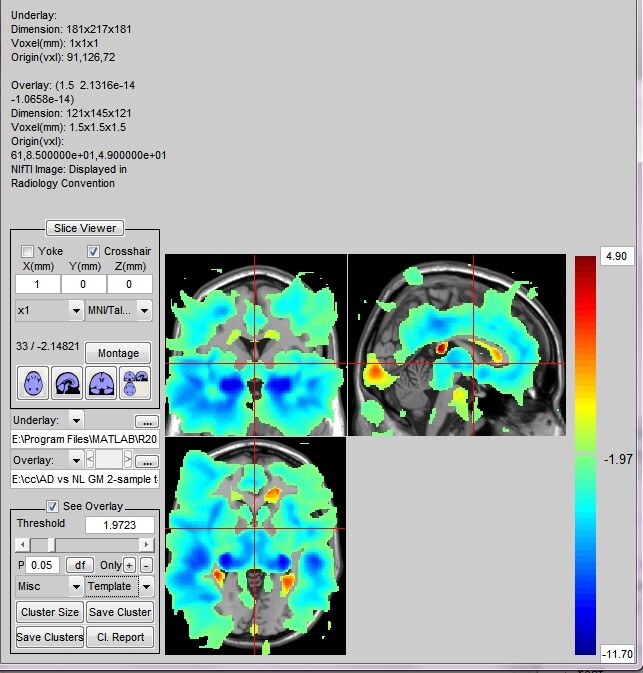
问题4:然后我根据视频里的教程,将cluster size第一个设定为54,第三个就是SPM_Criterion,点击Cl.Report,出现了report结果,但是问题是,如果我就是这样正负激活混合的话,报告出来有22个cluster,如果我单一的点击only+就会报告有18个cluster,only-就是7个cluster,这些cluster有重复的,但是也有不重复的,我很纳闷,难道不该是only+和only-的总和个数等于那个22么?
问题5:我点击了only+,only-以及直接点击save cluters,保存下来的roi一样吗?我看大小好像是一样的,但是接下来我利用这个roi.nii来针对AD的那些smwrp1*.nii处理计算所得文件的数量不同,比如我先是点击了only+再save的roi拿来处理得到了两万多个.mat和.txt文件,但是我要是用直接save的那个,处理了一天了八万多个.mat和.txt还没处理完????
问题6:通过extract ROI signals得到的结果,那些.mat和.txt文件到底是什么呢,就是比如txt文件里的一长列数值,比如2.3454575687E-4这样的,到底是什么意思呢?
恳请各位大大给予解答orz小白现在完全一头雾水,不知道该怎么办了QAQ

Submitted by ZHANG_RESTadmin on Sat, 12/20/2014 - 17:14 Permalink
Re: ...
你不是已经用REST sliceviewer打开双样本t检验的结果了么?这就行了啊。
FDR校正选择全脑模板,就是REST文件夹,mask里面的brainmask05 61 73 61 那个,选择AAL就错了。
超出范围的结果是因为你双样本t检验时没有给mask,给brainmask05 61 73 61 就好了。
cluster size第一个设定为54?为何?这里你不是FDR校正么?和cluster size没有关系。cluster size在视频里是做Alphasim校正用的。
report以正负混合的结果为准。
你要点save cluster一个一个保存ROI,用binary ROI做后续的Extract ROI signal,提取完会生成很多结果,要的是ROIsignal.txt,把这个打开就是每个被试这个ROI内部平均的信号值。
2.3454575687E-4是科学计数法,具体百度之。
Submitted by ilovec on Tue, 03/10/2015 - 10:29 Permalink
Re: ...
非常感谢老师的解答orz
然后我出现了一点新的小小的问题——
我用SPM做的双样本t检验,给的就是brainmask05那个,然后取的FDR校正,P值0.05
然后想要用rest计算ROI的体积,就是用的utilities的extract ROI signals,打开以后,选用的模板也是brainmask05,结果缩小了很多,成了这个样子,而且弹出了警示窗口——
问题1:
警示窗口如下:
我已经用的是这个模板了,怎么会出现这样的不匹配呢?
问题2:
之前您问我为何还要做cluster size,是因为我不管这个的话,不论是点击save cluster,还是report,都会弹出这个对话框
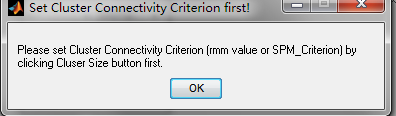
问题3:
我已经在SPM中做过FDR校正了,请问还需要在REST的那个图中,输入 P 0.05吗?如图所示
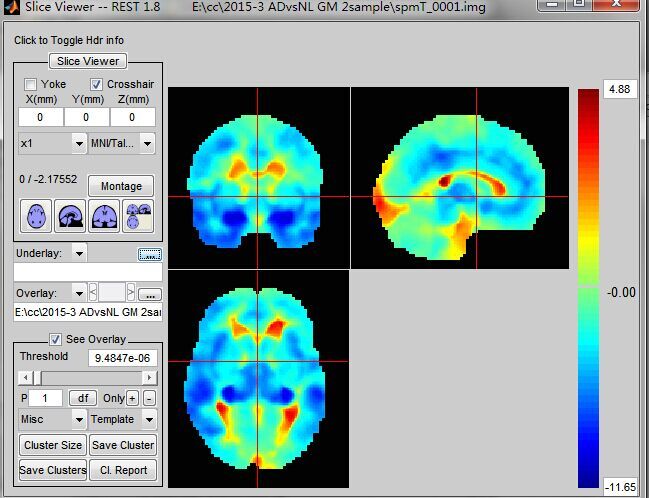
就是这个:
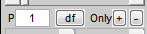
我已经在SPM的result那一步中进行到了FDR p0.05校正,现在如果我在这一栏p输入0.05,结果如下
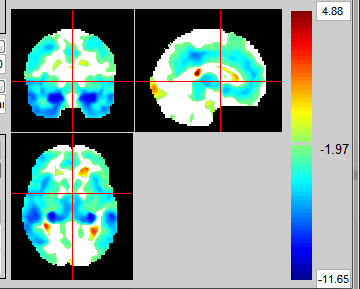
请问这样操作的结果是对的吗?