Submitted by Beginner on Tue, 10/28/2014 - 15:45
各位老师您好!
看您的有篇相关文献《Disrupted Causal Connectivity in Mesial Temporal Lobe Epilepsy》中做的voxel-wise Granger causality analysis,其中有用GCA计算结果与临床指标做相关分析,这里是用的 “Granger causality value ”与 disease duration 做的相关,我想问一下这里的相应脑区的“Granger causality value ”是怎么得到的?是相应脑区又与ROI做ROI-wise GCA计算得到的连接系数还是最初voxel-wise GCA计算统计结果提取出来的GCA值?

Submitted by jigongjun on Fri, 10/31/2014 - 14:21 Permalink
Re: voxel-wise GCA计算
Q1:相应脑区的“Granger causality value ”是怎么得到的?
A1:
1)voxel-wise GCA组间比较得到了一些差异脑区(e.g., region A and B...)
2) 可以用region A作为ROI,提取出每个人A与seed点的GCA值
3)用这些GCA值与duration做across subject的相关即可
Q2:“是相应脑区又与ROI做ROI-wise GCA计算得到的连接系数还是最初voxel-wise GCA计算统计结果提取出来的GCA值?”
A2: 是最初voxel-wise GCA计算统计结果提取出来的GCA值。
Submitted by Beginner on Mon, 11/03/2014 - 08:50 Permalink
Re: voxel-wise GCA计算
谢谢老师!十分感谢!可是老师我还有一个问题想请教您!
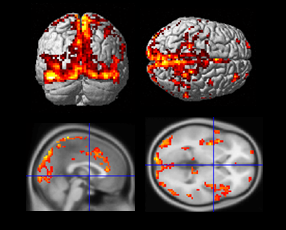 我觉得这个结果很不正常!不知道为什么!处理了好几遍都是这样!并且有y2x的统计结果是空白的,什么都没有!麻烦老师看到请给解答指点一下!老师辛苦!
我觉得这个结果很不正常!不知道为什么!处理了好几遍都是这样!并且有y2x的统计结果是空白的,什么都没有!麻烦老师看到请给解答指点一下!老师辛苦!
我处理了一批数据,我选择的是运动脑区中的M1做种子点进行voxel-wise GCA计算!为什么x2y的统计结果图中信号都出现在大脑两半球之间和大脑与小脑缝隙之间,图例:
Submitted by jigongjun on Tue, 11/04/2014 - 15:02 Permalink
Re: voxel-wise GCA计算
你的预处理步骤是?
用的系数GCA?
正常对照的数据?统计时,是否用了MASK?
Submitted by Beginner on Wed, 11/05/2014 - 11:51 Permalink
Re: voxel-wise GCA计算
老师您好!我的预处理步骤是去除前n个时间点的数据,时间层矫正,头动校正,T1分割空间标准化,平滑(4mm),去除协变量,去线性漂移,滤波。是不是不应该去除协变量?
我用的是系数GCA计算的。
正常对照的数据没有问题!统计时我选用了Brodmann_61x73x61的模板!
Submitted by jigongjun on Thu, 11/06/2014 - 09:39 Permalink
Re: voxel-wise GCA计算
把正常对照的图也贴上来看下吧(X2Y和Y2X),去除的协变量里包括全脑均值吗?
Submitted by Beginner on Thu, 11/06/2014 - 17:05 Permalink
Re: voxel-wise GCA计算
老师,您好!我上边贴的那张图就是正常对照组的x2y的统计结果!采用的Alphasim矫正(p=0.05,cluster=60),y2x的统计结果如果采用相同的阈值就是空白!全脑都没有信号!阈值设大一点就是如图结果!.png)
我的去除协变量里有去除全脑均值!老师全脑均值去和不去哪个更好一点呢?还是协变量本身就不用去呢?我看文献里有的去除有的没有去除!
而且老师我还想说一下!预处理时在DPARSF中有Detrend和Filter,REST-GCA中也有Detrend和Filter,这两个步骤在不同的里边做,最后计算统计出来的结果也不一样!不知道为什么?!
Submitted by jigongjun on Fri, 11/07/2014 - 19:02 Permalink
Re: voxel-wise GCA计算
去不去协变量(全脑均值),确实没有定论。你认同全脑均值可以反映一些生理噪音,就可以将它去除掉,否则就不去掉。
REST和dparsfa的Filter程序可能不太一样,不知道后来有没有统一。但是,最后的显著性结果不会有太大差异的,最多只是统计值略有不同。
Submitted by Beginner on Thu, 11/06/2014 - 22:52 Permalink
Re: voxel-wise GCA计算
多谢老师您的耐心解答!但是我还有疑惑的地方问一下老师,麻烦老师帮忙解答!
Q1:我想问一下系数GCA的结果的正负是代表positive and negative causal effects吗?看您的这个文献《Disrupted Causal Connectivity in Mesial Temporal Lobe
Epilepsy》结果图示中有写到!而且我也发现X2Y的正结果接近Y2X的负结果,X2Y的负结果接近Y2X的正结果。不知道作何解释?
Q2:voxel-wise GCA计算一般用系数GCA还是残差GCA计算比较好?我也有用残差GCA计算过,结算结果文件有GCA_NetFx2y,GCA_x2y,GCA_x2y_Transformed,GCA_y2x,GCA_y2x_Transformed,还有相应的以ZGCA开头的,不知道用哪种类型文件来做统计或者说具有统计意义?望老师指点!
Submitted by Zhou huiling on Sun, 07/02/2023 - 10:12 Permalink
Re: voxel-wise GCA计算
请问老师,这个做组间比较用的是spm的双样本t检验嘛