关于Deparsf处理DKI图像的问题
我在用Deparsf处理DKI的图像,做空间标准化,我想问下,是不是DKI图像也需要做Slicetiming等等步骤,还是直接空间标准化就好了?
- Read more about 关于Deparsf处理DKI图像的问题
- Log in or register to post comments
- 3222 reads
我在用Deparsf处理DKI的图像,做空间标准化,我想问下,是不是DKI图像也需要做Slicetiming等等步骤,还是直接空间标准化就好了?
请教一下各位老师们
为什么我用Dprasf 预处理后图像在脑底部出现黑色条带(见附件),而用其他方法预处理则没有出现?是否可以通过修改dparsf的处理过程得到改善,这样的结果对后面的分析会产生怎样的影响?
非常感谢!
各位老师,
请教一下,在常用的dparsf软件中做normalize 是用的什么方法?
非常感谢!
echo
呵呵,突然想到的,这样可行么?
做静息态磁共振1.5T和3.0T有什么区别?我看目前文献上大多是3.0T,如果我用1.5T的话是不是会有问题?多谢!
做静息态磁共振1.5T和3.0T有什么区别?我看目前文献上大多是3.0T,如果我用1.5T的话是不是会有问题?多谢!
各位老师好,
最近我用DPARSF得到了一个被试的空间标准化结果,扫描参数:
Sinmens,3.0T,GER-EPI,TE/TR=30/2000ms,flip angle = 90°,FOV = 1320 ×1320mm2, matrix = 64 × 64, slices = 33, slice thickness = 3 mm;没有用自身的T1像。得到下面的标准化结果,我想问问(1)这样的标准化结果是否可以采用,还是应该舍弃?(2)标准化结果是否与TE,TR有关?还是与头动相关?
各位老师,扣带回中部和后部(AAL 33-36) 到底是算在limbic network,还是算DMN?
做ROI—wise相关的时候,发现了和扣带回中后部的显著相关,这些到底算哪个网络部分可能大?
各位好! 最近正在常使用gretna的GUI进行网络分析,但输入参数必须是.mat吗,cell里包含多人的.mat文件不行吗?记得以前gretna最初版本的时候用代码是可以的。但现在GUI参数输入进去后,最是提示“The following file(s) are missing to process the pipeline :”,能成功处理的只有cell里的第一个mat文件,后面的都不行。
补充:
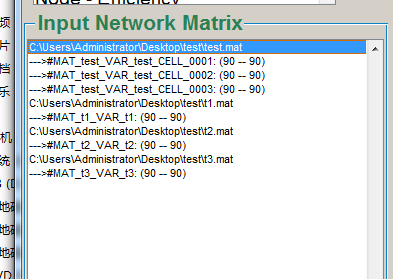
上面第一个就是我上传的cell格式的数据,它能读出来我三个矩阵,但处理的时候就只能处理第一个。下面是我把三个矩阵分别存成mat格式,再上传,就全部能正常处理。