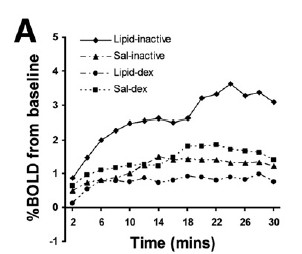
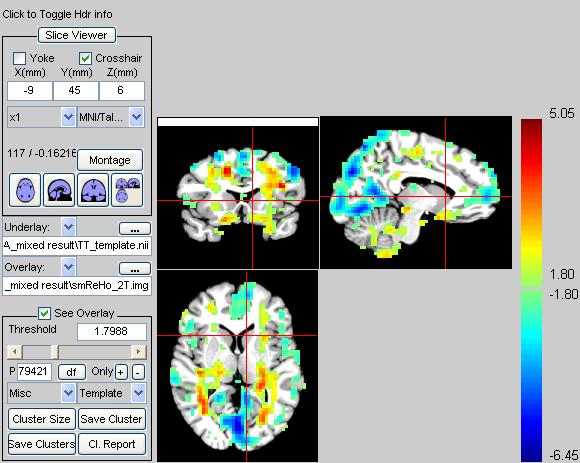
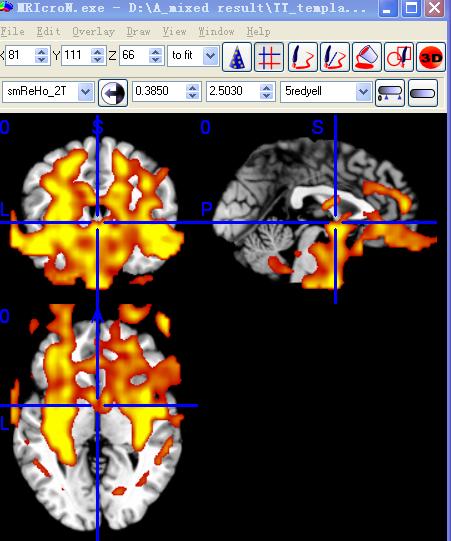
Questions in AlphaSim in REST
抱歉有两个不是问题的问题请教:
1. AFNI里AlphaSim还有关于user specified activate region情况,请问REST有考虑将来的版本升级呢?
另外请教,相应参数Zsep (separation between the mean of the noise distribution and the mean of the signal distribution)如何设定呢?
2. REST里AlphaSim关于xthr (xthr = sd*zthr + mean)计算,sd和mean都是每迭代一次,要累积前面迭代的值,我不是很明白。即如下:
count=count+nxyz; 为什么不每次迭代count都赋值为nxyz呢?
- Read more about Questions in AlphaSim in REST
- 2 comments
- Log in or register to post comments
- 62737 reads